pyvista.ImageDataFilters.median_smooth#
- ImageDataFilters.median_smooth(
- kernel_size=(3, 3, 3),
- scalars=None,
- preference='point',
- progress_bar: bool = False,
medianフィルターでデータを平滑化します.
各画素を,その画素を中心とした矩形近傍領域の中央値で置き換える中央値フィルタ.近傍領域は 3 次元以下でなければなりません.近傍領域の kernelSize の軸を 1 に設定すると,フィルターは 2 次元の中央値になります.
詳しくは vtkImageMedian3D を参照してください.
- パラメータ:
- kernel_sizesequence[
int], default: (3, 3, 3) (x_size, y_size, z_size)各次元におけるカーネルのサイズ(ボクセルの単位).デフォルトは3次元のメディアンフィルタ.2次元のメディアンフィルタを行いたい場合は,フィルタリングを行わない次元のサイズを 1 にセットします.- scalars
str,optional 処理に使用するスカラーの名前.デフォルトは現在アクティブなスカラーです.
- preference
str, default: "point" スカラーが指定されている場合,これはデータセット内で検索するために推奨される配列型です.
'point'または'cell'のいずれかでなければなりません.- progress_barbool, default:
False 進行状況を示す進行状況バーを表示します.
- kernel_sizesequence[
- 戻り値:
pyvista.ImageDataスカラーを平滑化した均一な格子.
警告
このフィルタをセルデータに適用すると,出力は同じ名前の新しいポイント配列に送られ,既存の同じ名前のポイントデータ配列は上書きされます.
例
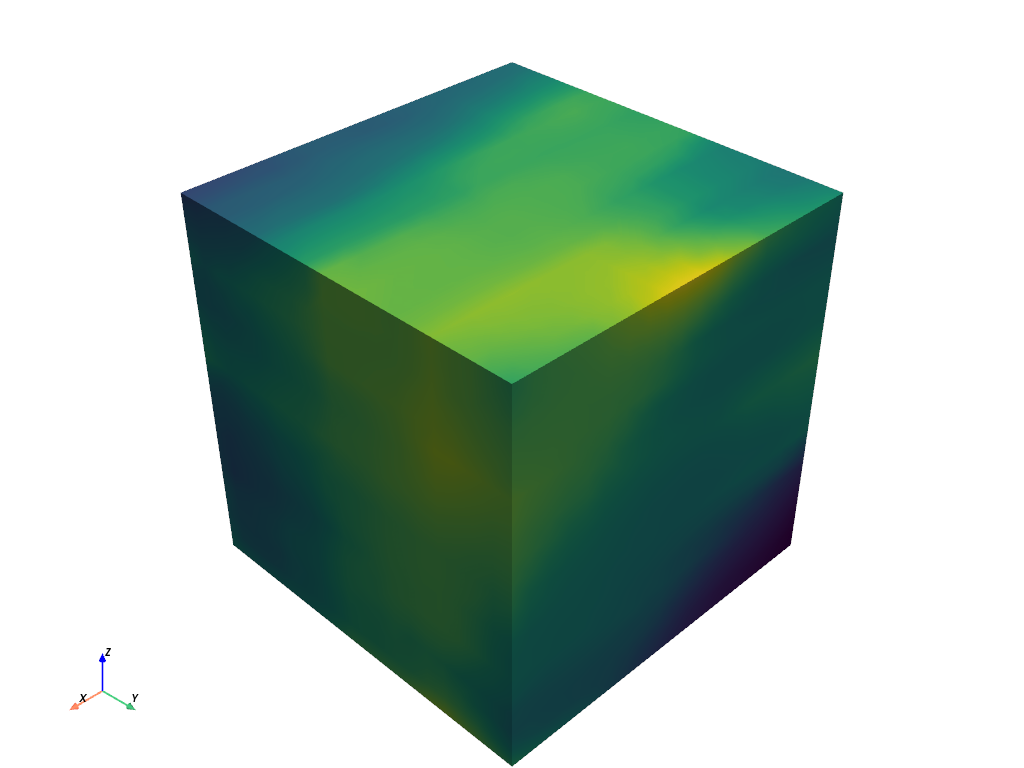
まず,スムージングするためのサンプルデータを作成します.ここでは
pyvista.perlin_noise()を用いて,意味のあるデータを作成します.>>> import numpy as np >>> import pyvista as pv >>> noise = pv.perlin_noise(0.1, (2, 5, 8), (0, 0, 0)) >>> grid = pv.sample_function(noise, [0, 1, 0, 1, 0, 1], dim=(20, 20, 20)) >>> grid.plot(show_scalar_bar=False)
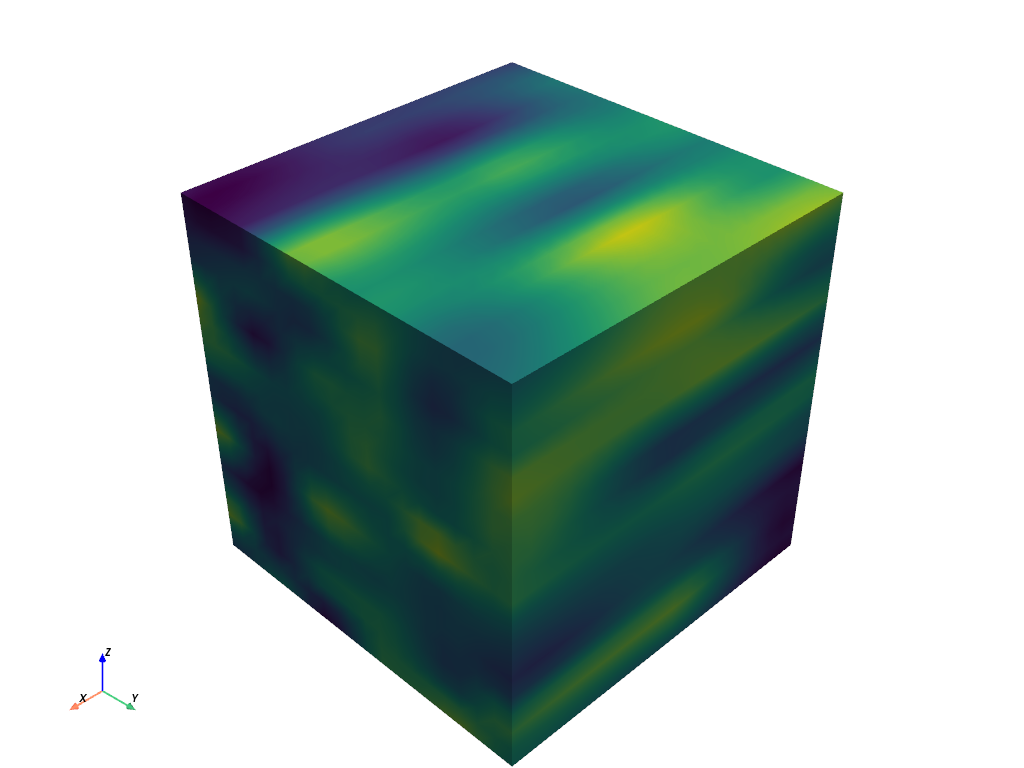
次に,サンプルデータを平滑化します.
>>> smoothed = grid.median_smooth(kernel_size=(10, 10, 10)) >>> smoothed.plot(show_scalar_bar=False)